Projet de consortium DevOCGen
Développement et applications de nouveaux outils pour la gestion et la conservation des populations naturelles à partir de données génomiques.
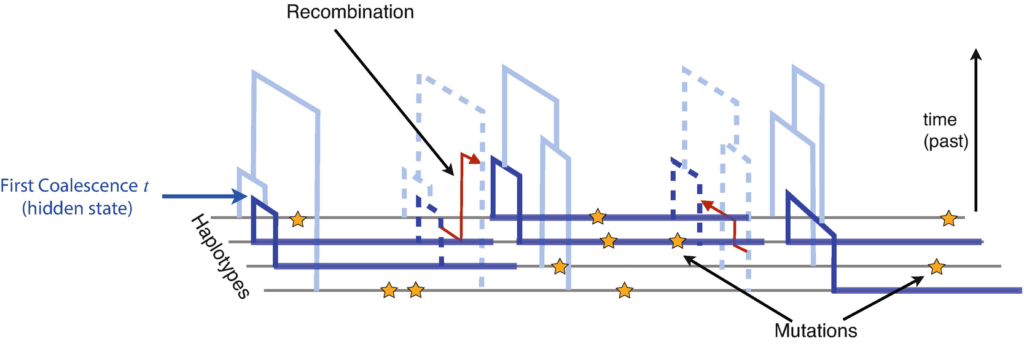
La gestion des populations menacées nécessite une connaissance fine de l’état démographique et génétique de ces populations : effectifs, fragmentation, consanguinité … Reconstruire la dynamique récente de ces différents paramètres est en particulier capital pour identifier les populations les plus vulnérables et les meilleures actions à mettre en place. Dans ce contexte, le polymorphisme génétique des populations représente une source d’information majeure mais encore peu utilisée en pratique.
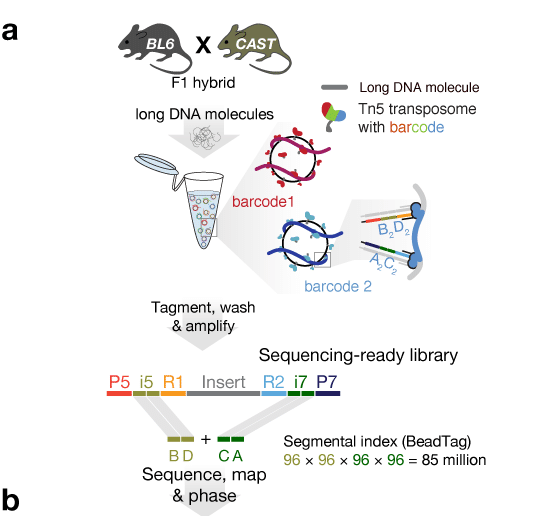
Le projet DevOCGen vise à optimiser cette utilisation en développant (i) une technologie de séquençage permettant d’obtenir de grands échantillons de génomes entiers pour un coût bien inférieur à celui des technologies actuelles et (ii) des méthodes statistiques permettant d’estimer la dynamique récente et locale des populations à partir de tels échantillons.
Ces outils seront appliqués à une série d’espèces vivant dans des habitats diversifiés et représentant des enjeux de conservation en Occitanie.





Partenariat
Le consortium constitué mobilisera différentes disciplines liées à la biologie moléculaire, l’écologie et l’évolution (génétique des populations en premier lieu, mais aussi génétique quantitative ou démographie), aux mathématiques et à l’informatique. Il couvrira un large spectre de compétences des plus théoriques (probabilités et statistique) aux plus technologiques (nouvelles approches de séquençage d’ADN), avec également une forte composante empirique (suivi à long-terme des populations sur le terrain).
Porteurs et co-porteurs du projet
| Porteurs | Simon BOITARD & Raphaël LEBLOIS, INRAE, CBGP |
| Co-porteur | Pierre-Alexandre GAGNAIRE, CNRS, ISEM |
| Co-porteur | Lounès CHIKHI, CNRS, CRBE |
| Co-porteur | Pierre-André CROCHET, CNRS, CEFE |
| Co-porteur | Simon BLANCHET, CNRS, SETE |
| Co-porteur | Joris BERTRAND, Université de Perpignan, LGDP |
Partenaires extérieurs
| Partenaire extérieur 1 | Bertrand SERVIN, INRAE, GenPhySE |
| Partenaire extérieur 2 | Jean-Michel MARIN, Muse, IMAG |
| Partenaire extérieur 3 | Olivier MAZET, INSA Toulouse, IMT |
| Partenaire extérieur 4 | Pierre DE VILLEMEREUIL, MNHN, ISYEB |
| Partenaire extérieur 5 | Jean-François LE GAILLARD, CNRS, IEES |
| Partenaire extérieur 6 | Gilles POTTIER, Nature en Occitanie (CEN) |
| Partenaire extérieur 7 | Hugues PARRINELLO, Plate-forme de Génomique MGX |
| Partenaire extérieur 8 | Erick DESMARAIS, Plate-forme de Génomique GenSeq |
| Partenaire extérieur 9 | Stéphane CAUET, Plate-forme de Génomique CNRGV |
| Partenaire extérieur 10 | Olivier BOUCHEZ et Céline LOPEZ-ROQUES, Plate-forme de Génomique GeT- PlaGe |
Résultats du projet DevOCGen
Les résultats du projet ont été présentés en décembre 2025 lors d’une conférence de restitution, dont les supports sont disponibles ici.
Publications
Arredondo, A., Corujo, J., Noûs, C., Boitard, S., Chikhi, L., & Mazet, O. (2025). Exact calculation of the expected SFS in structured populations. Theoretical Population Biology, 163, 50-61. https://doi.org/10.1016/j.tpb.2025.03.003
Jouniaux A., Arredondo A., Boitard S., Chikhi L., & Mazet O. (2025). Extending the IICR to complex non-stationary structured models. Genetics, sous presse. https://www.biorxiv.org/content/10.1101/2024.10.21.619462v1
Hobolt A., S. Boitard, A. Futschik, R. Leblois (2024). A matrix-analytical sampling formula for time-homogeneous coalescent processes under the infinite sites mutation model. Theoretical Population Biology, volume 163, 62-79. https://doi.org/10.1016/j.tpb.2025.03.002
Meyer, L., Barry, P., Riquet, F., Foote, A., Der Sarkissian, C., Cunha, R. L., … & Gagnaire, P. A. (2024). Divergence and gene flow history at two large chromosomal inversions underlying ecotype differentiation in the long‐snouted seahorse. Molecular Ecology, https://doi.org/10.1111/mec.17277
Muller T, Gautier M, Lombaert E, Leblois R, Sauné L, Branco M, Kerdelhué C, Perrier C. (2025). Population Genomics of Incipient Allochronic Divergence in the Pine Processionary Moth. Molecular Ecology e70189. https://doi.org/10.1111/mec.70189
Parreira B, Gpalakrishnan S, Chikhi, L (2025) Effects of social structure on effective population size change estimates. Evolutionary Applications; 18:e70063 https://doi.org/10.1111/eva.70063.
Romieu J, Camarata G, Crochet P-A, de Navascués M, Leblois R, Rousset F. (2025) Performance evaluation of adaptive introgression classification methods. Peer Community Journal, Volume 5, article no. e95. https://doi.org/10.24072/pcjournal.617
Tournebize R, and Chikhi L (2024) Ignoring population structure in hominin evolutionary models can lead to the inference of spurious admixture events. Nat Ecol Evol. https://doi.org/10.1038/s41559-024-02591-6
Uhl, M., de Navascués, M., Boitard, S., & Servin, B. (2024). SelNeTime: a python package inferring effective population size and selection intensity from genomic time series data. bioRxiv, https://doi.org/10.1101/2024.11.06.622284
Vishwakarma, R., Sgarlata, G.M., Soriano-Paños, D., Rasteiro, R., Maié, T., Paixão, T., Tournebize, R. and Chikhi, L. (2024), Species-Specific Traits Shape Genetic Diversity During an Expansion–Contraction Cycle and Bias Demographic History Reconstruction. Molecular Ecology, 34: e17597. https://doi.org/10.1111/mec.17597
Preprint :
Chikhi L, Rodriguez W, Paris C, Ha-Shan M, Jouniaux A, Arredondo A, Noûs C, Grusea S, Corujo JM, Lourenço I, Boitard S, Mazet O (2024). Extending the IICR to multiple genomes and identification of limitations of some demographic inferential methods. bioRxiv. https://doi.org/10.1101/2024.08.16.608273
Ha-Shan M, Thébaud, C, Tournebize, R, Gabrielli, M, Arredondo, A, Mila, B, Chikhi L (2024) Comparative population genomics of island birds reveal contrasting signals of ancient and recent population structure and panmixia. ResearchSquare, https://doi.org/10.21203/rs.3.rs-5290559/v1
Keilani H S, Fiston-Lavier A-S, Caminade P, Loiseau A, Galan M, Parrinello H, Brouat C, Leblois R, Avenant N, Pillay N, Ganem G, Smadja C M.Role of aridity in shaping adaptive genomic divergence and population connectivity in a Southern African rodent.bioRxiv 2025.07.29.667317; https://doi.org/10.1101/2025.07.29.667317
Rousset F, Leblois R, Estoup A, Marin J-M. 2025. A new iterative framework for simulation-based population genetic inference with improved coverage properties of confidence intervals. bioRxiv 2024.09.30.615940. https://doi.org/10.1101/2024.09.30.615940
Steux C, Couloigner, C, Arredondo, A, Rodríguez, WV, Mazet, O, Tournebize, R, Chikhi, L, (2024). On the demographic history of chimpanzees and some consequences of integrating population structure in chimpanzees and other great apes. bioRxiv, (2025) HPGG, doi: https://doi.org/10.1101/2024.06.14.599042